環状 RNA (circular RNA; circRNA)は、ショウジョウバエ、マウス、ヒトなどの動物細胞で見つかっている。植物細胞でも circRNA が見つかっているという報告例もある。その機能について、まだ解き明かされていない。しかし、一部の報告によれば、circRNA は miRNA に結合し、miRNA の働きを阻害することによって、遺伝子発現の転写後調節に関与している可能性がある。
circRNA の合成
circRNA は、直鎖 pre-mRNA が成熟する過程において、通常のスプライシングが逆方向で行われる back splicing によって合成される(Chen et al., 2015)。スプライシングが普通に行われるならば、pre-mRNA からイントロンが取り除かれて、mRNA が形成される。しかし、back splicing することによって、1 つあるいは複数のエキソンが 1 つの環を形成してしまうことがある。これが circRNA である。
ヒトの circRNA は一般的に 2、3 個のエキソンを含むことが報告されている。その長さは、一般的に数百塩基であるが、中には 4 千塩基前後のものも確認されている。また、1 つの遺伝子が選択スプライシングすることによって複数の転写物を生成できるように、1 つの遺伝子から複数の circRNA が作られる。1 つの遺伝子から最多で十数個の circRNA が作られることが確認されているが、ほとんどの場合 1、2 個である。最近のゲノムワイドな解析により、circRNA の発現量は、mRNA の発現量の 2-4% であると予想されている(Szabo et al., 2016)。
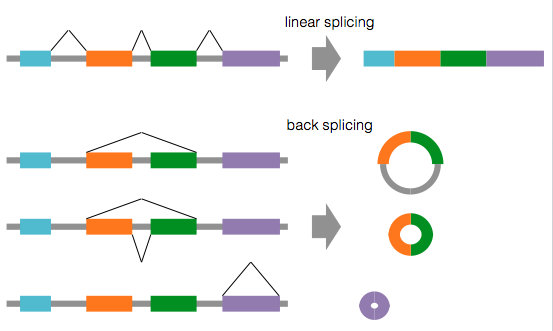
back splicing が行われるメカニズムはいくつか報告されている。
- スプライソソームが関与している circRNA 合成経路においては、U1 snRNA および U2 snRNP などの相互作用により back splicing が行われる。
- スプライソソームが関与していない合成経路においては、mRNA 前駆体の配列の一部(30-40 nt)が相補鎖を形成し、相補鎖となった部分が切断されて circRNA が生成される。
- RNA binding protein (RBP) が関与している circRNA 合成経路もある。muscleblind 遺伝子からタンパク質が作られ、このタンパク質が自身の mRNA 前駆体に結合することで、back splicing を引き起こし、circRNA の形成を促進する。このように、muscleblind 遺伝子から作られる circRNA は、それ自身のタンパク質が発現調節している。
circRNA の発現と機能
circRNA は一般的に発現が低いことが知られている。しかし、少数の遺伝子領域から作られる circRNA の量は、その遺伝子の直鎖 mRNA によりも多いことも報告されている(Salzman et al., 2013)。また、細胞の種類や組織特異的に発現量が高くなる circRNA も報告されている(Liang et al., 2014, Starke et al., 2015)。RNA-Seq を利用した研究では、哺乳類の脳に多くの circRNA が蓄積されていることが明らかになった。特に、神経が形成されるときに circRNA の発現量が高く、場合によってその遺伝子の直鎖 RNA の発現量よりも高い。
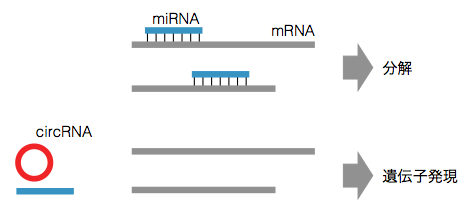
circRNA の機能については未知な部分が多い。その中で、よく知られている circRNA である ciRS-7 は miRNA スポンジとして機能することがわかっている。ciRS-7 が miR-7 のスポンジとしてよく知られ、ヒトやマウスの脳で発現する。細胞質において、ciRS-7 は miR-7 と結合することで、miR-7 が miR-7 結合型 mRNA に結合することを阻害する。そのため、ciRS-7 の発現が低下すると、miR-7 が miR-7 結合型 mRNA と結合するようになり、miR-7 結合型 mRNA の分解が進む。しかし、研究が進むにつれ、miRNA 標的サイトを持たない circRNA も見つかっている。そのため、circRNA は miRNA のスポンジとして機能する以外にも、何らかの機能を持つことが推測される。
circRNA と RNA-Seq
circRNA は環状 RNA であり、ポリ A テールを持たないのが特徴である。そのため RNA-Seq により circRNA の発現量を定量する場合、ライブラリー作成のときに、ポリ A テールを持つ RNA を選択的に取り除けばよい。具体的に、サンプルから RNA ライブラリーを精製し、次に、rRNA を選択的に分解する。さらに、RNase R 酵素を加えて、ポリ A テールを持つ直鎖 RNA を選択的に分解する。このように作成されたライブラリーには多くの circRNA が含まれていると考えられる。
circRNA を検出するためのソフトウェアとして circExplorer、circRNA_finder、CIRI、KNIFE などが開発されている。これらのソフトウェアは作られてまもないために、あるソフトウェアが検出した circRNA が、ほかのソフトウェアでは検出できなかったりすることがある(Hansen et al., 2016)。
circRNA のデータベース
circRNA のデータベースは、次の表にあげられたようにいくつか存在する。circRNA のデータベースで公開されているデータは、ほとんどヒトとマウスのデータからなる。また、データ量も、lncRNA や miRNA のデータベースに比べれば、少ない。
| データベース | 生物種 | データベースの特徴 |
| circBase | ヒト、マウス、線虫、ラティメリア | RNA-Seq のデータをスクリーニングして得られた circRNA のデータが保存されている。他のデータベースに比べて、生物種が多い。 |
| circNet | ヒト | RNA-Seq のデータセットを利用して構築された ceRNA の制御ネットワークが保存されている。circRNA miRNA および遺伝子間の相互作用を検索することが可能。 |
| circRNADb | ||
| deepBase | ヒト、マウス | RNA-Seq のデータをスクリーニングして得られた circRNA のデータが保存されている。circRNA 以外にも lncRNA や miRNA などの ncRNA のデータが保存されている。circRNA 以外の ncRNA に関しては、発現量や機能アノテーションなども調べることができる。 |
| plantcircbase |
References
- Detecting circular RNAs: bioinformatic and experimental challenges. Nat Rev Genet. 2016, 17(11):679-692. DOI: 10.1038/nrg.2016.114 PMID: 27739534
- Regulation of circRNA biogenesis. RNA Biol. 2015, 12(4):381-8. DOI: 10.1080/15476286.2015.1020271 PMID: 25746834
- Short intronic repeat sequences facilitate circular RNA production. Genes Dev. 2014, 28(20):2233-47. DOI: 10.1101/gad.251926.114 PMID: 25281217
- Exon circularization requires canonical splice signals. Cell Rep. 2015, 10(1):103-11. DOI: 10.1016/j.celrep.2014.12.002 PMID: 25543144
- Cell-type specific features of circular RNA expression. PLoS Genet. 2013, 9(9):e1003777. DOI: 10.1371/journal.pgen.1003777 PMID: 24039610
- Comparison of circular RNA prediction tools. Nucleic Acids Res. 2016, 44(6):e58. DOI: 10.1093/nar/gkv1458 PMID: 26657634